
Yanomami-Mutter mit Kind. © Cmacauley. CC BY-SA 3.0
Das Mikrobiom, die Bakterien, die auf und in uns leben, sind ein wesentlicher Faktor für unsere Gesundheit. Vergleiche zwischen den Mikrobiomen von Menschen, die noch ein ursprüngliches Jäger- und Sammler-Leben führen mit denen aus der westlichen Zivilisation können Aufschluss darüber geben, wie unsere natürliche Bakterienflora einmal ausgesehen haben könnte. Wissenschaftler hatten nun die Gelegenheit das Mikrobiom von Mitgliedern eines noch isoliert lebenden Stammes der Yanomami in Amazonien zu untersuchen. Wie sich herausstellte verfügen diese Menschen über eine größere Vielfalt an Darmbakterien, als Mitglieder der westlichen Zivilisationen. Darüber hinaus besitzt die Darmflora der Yanomami bereits eine Vielzahl potentieller Resistenzgene gegen Antibiotika, obwohl der Stamm bisher noch nie mit westlicher Medizin in Kontakt gekommen ist.
Maria Dominguez-Bello von der New York University School of Medicine in New York City, setzte gleich, nachdem sie mitbekommen hatte, dass in den Bergen Südvenezuelas ein noch isoliert lebender Yanomami-Stamm entdeckt worden war, alle Hebel in Bewegung, um das Mikrobiom dieser Menschen untersuchen zu dürfen. Isoliert lebende Stämme sind ein Glücksfall für die Mikrobiologen, denn ihre Bakterienflora ist noch nicht durch den Einsatz von Antibiotika und die westliche Ernährungsweise verändert. Von ihnen hoffen die Forscher zu lernen, welche Bakterien uns im Laufe unseres Zivilisationsprozesses verloren gegangen sind.
Mitarbeiter des Yanomami-Gesundheitsdienstes sammelten 2009 bei einem Besuch des Yanomami-Dorfes für die Wissenschaftler Bakterienproben aus der Mundhöhle, von der Haut und vom Stuhl von 34 der damals 54 Yanomami. Wie die Mitarbeiter des Gesundheitsdienstes feststellten beherbergten die Yanomami zwar viele Parasiten, waren aber dennoch gesund und litten nicht unter Zivilisationskrankheiten, wie Autoimmunerkrankungen, Diabetes, hohem Blutdruck oder Herzerkrankungen.
Große Artenvielfalt im Mikrobiom der Yanomami
Aufgrund bürokratischer Hindernisse bei der Überführung der Bakterienproben nach Amerika und der Zerstörung des Labors von Maria Dominguez-Bello durch den Hurricane Sandy verzögerte sich die Analyse der Proben um circa drei Jahre. Danach konnte die Forscherin mit ihrem Team endlich die Zusammensetzung des Mikrobioms der Yanomami untersuchen. Dazu sequenzierten sie die ribosomale RNA der Bakterien und identifizierten die jeweiligen Arten durch einen Abgleich mit Datenbankeinträgen. Die Zusammensetzung der Darmflora der Yanomami verglichen sie dann mit denen von Amerikanern, in Venezuela lebenden Guahibo-Indianern und Bauern aus Malawi. Dabei erwies sich die Darmflora der Yanomami als wesentlich artenreicher, als die der anderen Gruppen. Sie zeichnete sich unter anderem durch eine große Zahl an Prevotella, Helicobacter, Oxalobacter, und Spirochaeta–Arten aus. Bei Menschen, die in der westlichen Zivilisation leben kommen diese Arten kaum oder gar nicht vor.
Das Microbiom und unsere Gesundheit
Forscher, die unser Mikrobiom untersuchen, vermuten schon lange, dass so manche Zivilisationskrankheit mit einem Fehlen bestimmter Darmbakterien einher geht. Oxalobacter etwa soll die Bildung von Nierensteinen verhindern helfen. Die Erforschung kausaler Zusammenhänge zwischen der Ab- oder Anwesenheit einer Bakterienart und dem Auftreten einer Krankheit gestaltet sich jedoch schwierig, da die Forschung sich oft darauf beschränkt positive oder negative Korrelationen aufzustellen. Diese erlauben jedoch naturgemäß nicht einen Ursache-Wirkungs-Nachweis. Denn bei einer reinen Korrelation kann entweder der Zufall im Spiel sein, oder eine Krankheit kann der Grund dafür sein, dass sich die Darmflora geändert hat und nicht umgekehrt die Darmflora der Grund für die Kranḱheit. Unsere Darmflora ist darüber hinaus stark von dem abhängig was wir zu uns nehmen. So ist sie etwa davon abhängig, ob wir viele Vollkornprodukte oder hauptsächlich Weißmehl oder viel oder weniger Fett zu uns nehmen (Literatur siehe unten). Auch die Zusammensetzung der Darmflora der Yanomami könnte, wie die Forscher zugeben, von den saisonal gerade verzehrten Nahrungsmitteln abhängen und damit variabel sein.
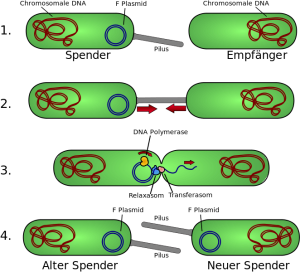
Schematische Zeichnung bakteriellen Gentransfers:
1. Spenderzelle produziert F-Pilus.
2. Pilus verbindet Zellen.
3. Einzelstrang des Plasmids wird übertragen
4. Beide Zellen produzieren einen Sexpilus und sind jetzt mögliche Spender.
© Matthias M.. CC BY-SA 3.0
Yanomami und die Antibiotikaresistenzen
Das Team um Gautam Dantas von der Washington University in St. Louis durchsuchte die Proben der Darm- und Mundflora der Yanomami nach Genen, die Antibiotikaresistenzen vermitteln. Und wurden fündig: gleich 28 Gene identifizierten sie, die die Bakterien vor Antibiotika schützen könnten, wenn sie diesen denn ausgesetzt wären. Wobei 14% dieser Gene bereits über Möglichkeiten für den Austausch dieser Gene zwischen verschiedenen Bakterienarten verfügten: Sie befanden sich auf Plasmiden, die leicht zwischen Bakterien ausgetauscht werden können oder waren von Transposons flankiert, die es den Genen erlauben innerhalb des Bakteriengenoms zu springen oder sich auf einem Plasmid einzufügen. Die von den Forschern bei den Yanomami entdeckten Resistenzgene waren oft fast identisch mit Resistenzgenen, die bei anderen Darmbakterien in der westlichen Zivilisation verbreitet sind. Einige dieser Gene wirkten sogar gegen neueste synthetische Antibiotika.
Antibiotikaresistenzen sind ein uraltes Phänomen
Das Auftreten dieser Resistenzen erklären sich die Forscher damit, dass die Darmbakterien der Yanomami ein breites Repertoire an Wirkstoffen entwickelt haben, mit dem sie sich verschiedenster Giftstoffe erwehren können, die ihre Existenz bedrohen. Zu ähnlichen Schlüssen sind bereits andere Studien gekommen, die Antibiotikaresistenzen bei isoliert lebenden Amazonasstämmen nachgewiesen haben (Literatur siehe unten). Andere Forscher haben sogar Resistenzmechanismen gegen Antibiotika in Bakterien aus 5000 Jahre altem Permafrostboden entdeckt (siehe Literatur weiter unten). Es scheint also in der Natur bereits seit Langem eine Vielzahl von Substanzen zu geben, die das Wachstum von Bakterien bedrohen und gegen die sich diese daher durch die Bildung geeigneter Gegenmittel schützen.
von Ute Keck
Originalartikel:
Jose C. Clemente, Erica C. Pehrsson, Martin J. Blaser, Kuldip Sandhu, Zhan Gao, Bin Wang, Magda Magris, Glida Hidalgo, Monica Contreras, Óscar Noya-Alarcón, Orlana Lander, Jeremy McDonald, Mike Cox, Jens Walter, Phaik Lyn Oh, Jean F. Ruiz, Selena Rodriguez, Nan Shen, Se Jin Song, Jessica Metcalf, Rob Knight, Gautam Dantas, M. Gloria Dominguez-Bello. The microbiome of uncontacted Amerindians. Science Advances 17 Apr 2015: Vol. 1 no. 3 e1500183. DOI: 10.1126/sciadv.1500183
Weitere Literatur zu Antibiotikaresistenzen bei isoliert lebenden Amazonasstämmen:
Bartoloni A, Pallecchi L, Rodríguez H, Fernandez C, Mantella A, Bartalesi F, Strohmeyer M, Kristiansson C, Gotuzzo E, Paradisi F, Rossolini GM. Antibiotic resistance in a very remote Amazonas community. Int J Antimicrob Agents. 2009 Feb;33(2):125-9. doi: 10.1016/j.ijantimicag.2008.07.029. Epub 2008 Oct 22.
Pallecchi L, Riccobono E, Mantella A, Fernandez C, Bartalesi F, Rodriguez H, Gotuzzo E, Bartoloni A, Rossolini GM. Small qnrB-harbouring ColE-like plasmids widespread in commensal enterobacteria from a remote Amazonas population not exposed to antibiotics. J Antimicrob Chemother. 2011 May;66(5):1176-8. doi: 10.1093/jac/dkr026. Epub 2011 Feb 7.
Literatur zu Antibiotikaresistenzen, die aus Bakterien in arktischen Bodenproben isoliert wurden:
Perron GG1, Whyte L2, Turnbaugh PJ3, Goordial J2, Hanage WP4, Dantas G5, Desai MM6. Functional characterization of bacteria isolated from ancient arctic soil exposes diverse resistance mechanisms to modern antibiotics. PLoS One. 2015 Mar 25;10(3):e0069533. doi: 10.1371/journal.pone.0069533. eCollection 2015.
Literatur zur Variation der Darmflora in Abhängigkeit von der Ernährung:
Zhou AL1, Hergert N1, Rompato G2, Lefevre M3. Whole grain oats improve insulin sensitivity and plasma cholesterol profile and modify gut microbiota composition in C57BL/6J mice. J Nutr. 2015 Feb;145(2):222-30. doi: Link zum Artikel. Epub 2014 Dec 10.
Martínez I1, Muller CE, Walter J. Long-term temporal analysis of the human fecal microbiota revealed a stable core of dominant bacterial species. PLoS One. 2013 Jul 16;8(7):e69621. doi: Link zum freien Artikel. Print 2013.
Hildebrandt MA1, Hoffmann C, Sherrill-Mix SA, Keilbaugh SA, Hamady M, Chen YY, Knight R, Ahima RS, Bushman F, Wu GD. High-fat diet determines the composition of the murine gut microbiome independently of obesity. Gastroenterology. 2009 Nov;137(5):1716-24.e1-2. doi: Link zum freien Artikel. Epub 2009 Aug 23.

