Pseudomonas aeruginosa. © public domain.
Bakterien müssen sich ständig an wechselnde Umweltbedingungen anpassen, um zu überleben. Diese Herausforderung meistern sie, indem sie koordiniert bestimmte Gene aktivieren, die sie unter den jeweiligen Lebensbedingungen benötigen. Forscher haben nun gezeigt, dass sich Bakterien schneller und nachhaltiger an veränderte Umweltbedingungen anpassen können, wenn sie über die Fähigkeit verfügen, ganze Gengruppen koordiniert zu aktivieren. Das berichtet das Helmholtz Zentrum für Infektionsforschung.
Das Bakterium Pseudomonas aeruginosa ist einer der in Deutschland am weitesten verbreiteten Krankenhauskeime. Etwa 10% aller Krankenhausinfektionen gehen auf sein Konto. Dabei ist die Palette der von ihm ausgelösten Erkrankungen umfangreich: Er ist nicht nur dazu in der Lage eine Hämolyse, das Auflösen roter Blutkörperchen, auszulösen. Sondern er verfügt auch über ein Toxin, das Exotoxin A, das mit dem Diphetherietoxin verwandt ist und die Proteinsynthese in der befallenen Zelle zum Erliegen bringt. Er kann Lungenentzündungen bei immunsupprimierten Patienten, aber auch Harnwegsinfektionen, Darmerkrankungen, Hirnhautentzündungen und Entzündungen des äußeren Gehörganges verursachen. Damit nicht genug kann sich dieser Überlebenskünstler auch im Wasser oder Boden vermehren. Egal, wo er vorkommt, gelingt es ihm immer wieder sich extrem schnell an seine neue Umwelt anzupassen und selbst unter extremen Bedingungen zu überleben. Forscher am Helmholtz Zentrum für Infektionsforschung haben nun einen wichtigen Mechanismus aufgedeckt, der es diesem Erreger ermöglicht, sich schnell an neue Umweltbedingungen anzupassen.
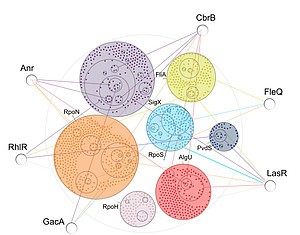
Modularer Aufbau des Genoms von Pseudomonas aeruginosa: Globale Transkriptionsfaktoren kombinieren verschiedene Sigmafaktoren zu funktionellen Gruppen. © HZI/Eckweiler
Dazu greift der Keim auf spezielle Transkriptionsregulatoren seines Genoms, sogenannte Sigma-Faktoren, zurück. Diese Proteine spielen eine entscheidende Rolle bei der koordinierten Aktivierung ganzer Gruppen von Genen. Um sich an unterschiedliche Umweltbedingungen anpassen zu können verfügt das Bakterium über verschiedene Sigma-Faktoren, die es je nach Bedarf produziert. Diese ordnen die Gene verschiedenen Gruppen zu, die damit auf einfache Weise gleichzeitig aktiviert werden können. Befindet sich das Bakterium beispielsweise im Wasser, so bildet es einen anderen Satz von Sigma-Faktoren, als wenn es sich im Blut eines Patienten vermehrt. Global wirkende Transkriptionsfaktoren bündeln dann diese regulatorischen Einheiten: Sie sorgen dafür, dass alle von den vorliegenden Sigma-Faktoren zusammen gefassten Gengruppen tatsächlich aktiviert und die benötigten Proteine synthetisiert werden.
So können die Transkriptionsfaktoren auf die bereits vorhandene Sortierung durch die Sigma-Faktoren zurückgreifen, um die für veränderte Umweltbedingungen notwendigen Gene zu finden. Auf diese Weise müssen sie bei evolutionären Anpassungen nicht alle notwendigen Gene einzeln optimieren, sondern können sich bereits zusammengefasster Gengruppen bedienen, aus denen sie nur einen bestimmten Bausteine optimieren müssen. Dadurch kann die Evolution nicht nur schneller, sondern auch effektiver erfolgen. Denn es ist wesentlich einfacher und schneller, einen einzelnen Baustein zu optimieren, als das gesamte Genom verändern zu müssen. Und dieses Baukastenprinzip kommt nicht nur bei der Evolution von Bakterien zum Einsatz, sondern ist auch ein wesentlicher Motor der Evolution höherer Lebewesen.
von Ute Keck
Originalpublikation:
Sebastian C. Binder, Denitsa Eckweiler, Sebastian Schulz, Agata Bielecka, Tanja Nicolai, Raimo Franke, Susanne Häussler & Michael Meyer-Hermann. Functional modules of sigma factor regulons guarantee adaptability and evolvability. Scientific Reports. 2016 Feb 26. DOI: 10.1038/srep22212

