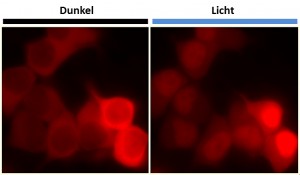
LINuS vermittelt die Licht-gesteuerte Wanderung des rot gefärbten Proteins in den Zellkern einer menschlichen Nierenzelle. © dkfz.de
Forscher der Universität Heidelberg und des Deutschen Krebsforschungszentrums (DKFZ) haben eine neue Methode entwickelt, mit der sie Prozesse in lebenden Zellen über Lichtsignale steuern können. Dieses System ermöglicht Studien über die Bewegung von Proteinen innerhalb der Zelle und ist daher sowohl für die Grundlagen- als auch für die angewandte Forschung interessant. Ihre Ergebnisse haben die Wissenschaftler jetzt in der Fachzeitschrift Nature Communications veröffentlicht. Gemeinsame Pressemitteilung von Deutsches Krebsforschungszentrum (DKFZ) und Universität Heidelberg.
Das neue System heißt wie der Gefährte des Zeichentrickhundes Snoopy: LINuS steht für „light-inducible nuclear localization signal“, also ein durch Licht induzierbares Signal, mit dem Proteine in den Zellkern gelenkt werden können. Die Besonderheit der neuen Methode ist, dass die Forscher über Licht direkt in die Prozesse lebender Zellen eingreifen können. Die Vorteile liegen klar auf der Hand: So, wie man Licht ein- und ausschalten kann, so kann auch LINuS ein- und wieder ausgeschaltet werden – und das gleich mehrmals hintereinander. Darüber hinaus hinterlässt es keine anderen Spuren in der Zelle, die wissenschaftliche Beobachtungen beeinflussen könnten. Gegenüber der Steuerung durch chemische Signale eröffnet LINuS zudem die Möglichkeit, gezielt einzelne Zellen aus einem Zellverband zu untersuchen. Da LINuS über eine kurze DNA-Sequenz an jedes beliebige Protein angehängt werden kann, stellt es ein universell einsetzbares Mittel dar, um verschiedenste Prozesse in Säuger- und Hefezellen zu untersuchen. Darüber hinaus lässt sich die Stärke des Signals über die Lichtintensität und die Dauer der Bestrahlung variieren. „Wir können verschiedene Versionen von LINuS sozusagen ,personalisiert‘ an die Bedürfnisse des untersuchten Proteins anpassen“, sagt der Leiter der Studie, Professor Roland Eils, der am Deutschen Krebsforschungszentrum und an der Universität Heidelberg forscht. „Das eröffnet uns eine Vielzahl von denkbaren Anwendungsgebieten.“
Das neue System basiert auf einem lichtempfindlichen Protein, das in Pflanzen an der Bewegung in Richtung des Sonnenlichts beteiligt ist. „Wir haben dieses pflanzliche Protein schrittweise in einen lichtabhängigen Protein-Shuttle-Service umgebaut, der sogar in menschlichen Zellen funktioniert“, sagt Dominik Niopek, der Erstautor der Studie. Das Signal, das den Transport des zugehörigen Proteins in den Zellkern vermittelt, ist im Dunkeln im LINuS-Anhänger (dem umgebauten Pflanzenprotein) „versteckt“ und daher ausgeschaltet. Erst bei Lichteinfall wird es freigelegt und bewirkt den Transport des markierten Proteins vom Zellplasma in den Zellkern. Ist dieses Protein beispielweise ein Transkriptionsfaktor, führt dies zum Anschalten bestimmter Gene und beeinflusst so direkt verschiedene Funktionen der Zelle. Getestet wurde das neue System zunächst mit einem fluoreszenten „Berichterstatter“: so konnten die Heidelberger Forscher die Wanderung des leuchtenden Proteins direkt unter dem Mikroskop beobachten. Die Wissenschaftler bewiesen aber auch, dass sie mit Hilfe von LINuS direkt in grundlegende, zelluläre Funktionen eingreifen können: so konnten sie – nur durch das Einschalten von Licht – bestimmte Gene anschalten und sogar Zellen in die Zellteilung treiben. In Krebszellen verläuft diese Zellteilung häufig schnell und ohne jegliche Kontrolle und führt zu genetischen Defekten, die das Tumorwachstum verstärken oder die Resistenz gegenüber bestimmten Medikamenten fördern. Außerdem sind in Krebszellen genetische Reparaturmechanismen häufig fehl- oder sogar ausgeschaltet. „All diesen Prozessen liegt eine komplexe, in gesunden Zellen wohlkoordinierte Bewegung der beteiligten Signalproteine zugrunde“, erklärt Barbara di Ventura, die die Arbeitsgruppe für Synthetische Biologie in der Abteilung von Roland Eils leitet. „Deren Erforschung steckt derzeit noch in den Kinderschuhen und kann nun mithilfe von LINuS vorangetrieben werden.“
Entscheidend für zukünftige Forschungsansätze sei insbesondere die Möglichkeit, die Aktivität bestimmter Gene räumlich und zeitlich zu kontrollieren, erklärt Dominik Niopek. „Es genügt nicht, ein Protein in einer Krebszelle einfach nur an- oder auszuschalten. Die Bewegung krebsrelevanter Proteine innerhalb der Zelle, wie beispielsweise die des Wächterproteins p53, ist ebenso wichtig. Diese können wir nun mit LINuS erforschen.“
Roland Eils und Barbara Di Ventura arbeiten seit Jahren in der Synthetischen Biologie. Das aufstrebende Forschungsfeld entwickelt Werkzeuge, um Zellen mit vollkommen neuen Eigenschaften auszustatten – und so neue Anwendungsgebiete in medizinischen, biotechnologischen und umweltrelevanten Bereichen zu erschließen. LINuS ist ein neues Werkzeug im Baukasten der Forscher. „Das Feld der Optogenetik, in der man Proteinaktivität mit Licht steuern kann, entwickelt sich zur Zeit rapide, weil Licht ein idealer, gezielter und nahezu nebenwirkungsfreier ,Schalter‘ ist, um Proteine in einzelnen Zellen zu steuern“, sagt Barbara Di Ventura. Im letzten Jahr holte das von Eils, Di Ventura und Niopek betreute studentische Team beim internationalen iGEM-Wettbewerb in Boston den Weltmeistertitel der Synthetischen Biologie nach Heidelberg.
Forschungsmeldung des Deutschen Krebsforschungszentrums (DKFZ) vom 21.07.2014.
Engineering light-inducible nuclear localization signals for precise spatiotemporal control of protein dynamics in living cells. Niopek D, Benzinger D, Roensch J, Draebing T, Wehler P, Eils R, Di Ventura B. Nat Commun. 2014 Jul 14;5:4404. doi: 10.1038/ncomms5404.

