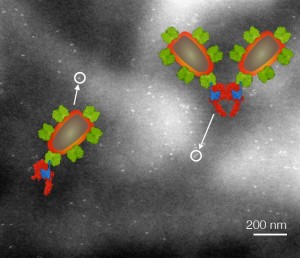
Die Auflösung der Liquid STEM Mikroskopie ermöglicht es, Einzelmoleküle und Dimere der HER2-Rezeptoren zu unterscheiden. © Leibniz Institut für neue Materialien
Mit einem neuen Mikroskopie-Verfahren haben Forscher die Verteilung bestimmter wachstumsfördernder Rezeptoren auf Brustkrebs-Zellen untersucht. Wie sie dabei entdeckten, fehlen einer kleinen Gruppe der Tumorzellen die krebsfördernden Rezeptor-Paare, sogenannte Dimere. Vermutlich handelt es sich bei diesen Zellen um ruhenden Zellen. Sie könnten laut den Wissenschaftlern der Grund für Resistenz und ein erneutes Tumorwachstum nach einer Antikörper-Therapie gegen die Rezeptoren sein.
Bei etwa einem Fünftel aller Brustkrebsfälle produzieren die Tumorzellen ungewöhnlich große Mengen eines bestimmten Rezeptors für Wachstumsfaktoren. Dieser so genannte HER2-Rezeptor sorgt für das ungehemmte Wachstum dieser Tumoren.
Antikörper, die gezielt an den HER2-Rezeptor binden („Herceptin“), sind ein häufig eingesetztes Medikament gegen diese Tumoren. Allerdings entwickeln etwa zwei Drittel der „HER2-positiven“ Tumorzellen Resistenzen gegen den Antikörper – die Gründe dafür waren bisher nicht bekannt.
Wissenschaftler aus dem Deutschen Krebsforschungszentrum und vom Leibniz-Institut für neue Materialien (Saarbrücken) untersuchten nun mit einem speziellen Mikroskopie-Verfahren die HER2-überexprimierende Brustkrebs-Zelllinie SKBR3. Dabei entdeckten sie eine kleine Population von Zellen, die keine HER2-Dimere auf ihrer Oberfläche trug.
Genau diese Dimere aus zwei aneinandergelagerten Rezeptormolekülen sind es aber, die das wachstumsfördernde Signal ins Zellinnere weiterleiten und Angriffspunkt der therapeutischen Antikörper sind. Aufgrund der Eigenschaften der Zellmembran vermuten die Forscher, dass es sich bei der Subpopulation um ruhende Zellen handelt, die Stammzell-Eigenschaften besitzen und daher für die Rückfälle nach einer Behandlung mit dem Antikörper-Medikament verantwortlich sein könnten.
Für ihre Analysen nutzten die Forscher ein als Liquid STEM bezeichnetes Mikroskopie-Verfahren, das Aufnahmen von intakten Zellen in ihrem flüssigen Medium ermöglicht. Mit dieser Methode untersuchten sie die Verteilung der HER2-Membranproteine auf den Zellen. Das Verfahren erlaubt, zwischen einzelnen Rezeptoren, Dimeren oder größeren Aggregaten zu unterscheiden.
HER2 gehört zur Gruppe der „epidermalen Wachstumsfaktor-Rezeptoren“ (EGF-R), die die Zellteilung anregen. Die übrigen Mitglieder dieser Rezeptor-Familie können sich erst dann zu Dimeren zusammenlagern, wenn ein Wachstumsfaktor an sie gebunden hat. HER2 dagegen braucht diesen Auslöser zur Paarbildung nicht und kann sich daher auch in Abwesenheit von Wachstumsfaktoren zusammenlagern und die Krebszelle zur Teilung anregen.
Das Antikörper-Medikament Herceptin verhindert die Zusammenlagerung zweier HER2-Moleküle zu einem Dimer. Außerdem markiert der Antikörper die Tumorzellen für das Immunsystem, so dass diese von „natürlichen Killerzellen“ des Immunsystems vernichtet werden können.
Die kleine, ruhende Zellpopulation, die keine HER2-Dimere auf ihrer Membran trägt, wird daher nicht für das Immunsystem kenntlich gemacht und könnte dadurch der Antikörper-Therapie entgehen.
Die entscheidende Zusammenlagerung zweier HER2-Molelüle war bislang meist mit biochemischen Methoden untersucht worden, die keinen Aufschluss über die einzelne Zelle geben konnten. Mit der Liquid STEM-Mikroskopie konnte nun erstmals das Verhalten der HER2-Moleküle auf lebenden, individuellen Krebszellen in Flüssigkeit beobachtet werden. Bei der herkömmlichen Elektronenmikroskopie dagegen müssen die Zellen entweder in Kunststoff eingebettet oder tiefgefroren werden.
Obwohl die HER-Proteine seit langem im Fokus der Krebsmediziner stehen, war die wichtige Tatsache, dass die HER2-Rezeptoren auf Krebszellen ungleich verteilt sein können, bislang nicht erkannt worden. Die jetzigen Ergebnisse sind der hohen räumlichen Auflösung der Liquid STEM Mikroskopie zu verdanken, mit der sich große Mengen intakter Zellen analysieren lassen.
Deutsches Krebsforschungszentrum (DKFZ), 20.07.2015
Originalpublikation:
D. B. Peckys, U. Korf, N. de Jonge: Local variations of HER2 dimerization in breast cancer cells discovered by correlative fluorescence and liquid electron microscopy. Science Advances, 2015, DOI: 10.1126/sciadv.1500165

