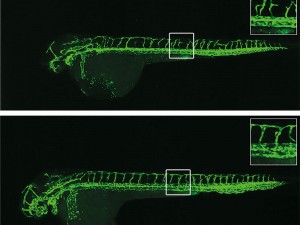
Blutgefäße einer Zebrafisch-Larve: Fehlt das Gen egfl7, bilden sich die Blutgefäße (grün gefärbt) nicht korrekt. In genetisch unveränderten Fischlarven mit egfl7 führt ein Wirkstoff, der egfl7 blockiert, zu starken Störungen des Gefäßwachstums (oben). In Fischlarven ohne egfl7-Gen kompensieren dagegen andere Gene den Ausfall, so dass der Hemmstoff das Wachstum der Blutgefäße kaum beeinträchtigt (unten).
© MPI f. Herz- und Lungenforschung
Neue Methoden zur Veränderung des Erbguts sind derzeit in aller Munde: Mit der CRISPR/Cas-Methode beispielsweise können Wissenschaftler Teile des genetischen Codes eines Gens ausschneiden und es dadurch ausschalten. Daneben gibt es auch Wege, die Übersetzung von Genen in Proteine zu verhindern. Beide Herangehensweisen verhindern die Bildung eines bestimmten Proteins und sollten vergleichbare Folgen für einen Organismus haben. Es hat sich jedoch herausgestellt, dass es unterschiedliche Konsequenzen haben kann, wenn ein Gen ausgeschaltet oder lediglich blockiert wird. Wissenschaftler haben nun herausgefunden, dass andere Gene für ein ausgeschaltetes Gen einspringen und die Auswirkungen abmildern oder sogar komplett ausgleichen können. Wird dagegen nur die Protein-Produktion blockiert, so scheint es nicht zu einer solchen Kompensation zu kommen. Diese neuen Erkenntnisse helfen sowohl dabei, die Ergebnisse molekularbiologischer Studien richtig zu deuten, als auch Gentherapien zur Behandlung von Krankheiten zu entwickeln.
Um die Funktion eines Gens unbekannter Funktion untersuchen zu können, schalten Wissenschaftler dieses oft aus und beobachten, welche Auswirkungen das auf den Organismus hat. Dabei können sie etwa mit Hilfe von Enzymen ein DNA-Stück aus dem Gen ausschneiden, so dass die genetische Information für ein funktionsfähiges Protein fehlt. Ein solches Verfahren wird als „Gen-Knock-out“ bezeichnet. Beim „Gen-Knock-down“ dagegen blockieren Wissenschaftler die Bildung eines Proteins mit besonderen Wirkstoffen, wie etwa sogenannten mikroRNAs.
Inzwischen hat sich jedoch herausgestellt, dass Studien mit Knock-out- und Knock-down-Tieren zu unterschiedlichen Ergebnissen führen können. Wissenschaftler aus der Arbeitsgruppe von Didier Stainier am Max-Planck-Institut für Herz- und Lungenforschung haben nun die Gründe dafür entschlüsselt. Dazu untersuchten die Forscher in Zebrafischen ein Gen namens egfl7. Dieses Gen ist an der Entstehung von Bindegewebe in der Wand von Blutgefäßen beteiligt und stabilisiert diese. Dadurch reguliert egfl7 das Gefäßwachstum.
Entwicklungsbiologen sind sich jedoch uneins, was im Körper des Fischs passiert, wenn das egfl7-Gen ausgeschaltet wird. „Wird das Gen mit einem Knock-down-Verfahren blockiert, entwickeln sich die Blutgefäße in den Fischlarven nicht korrekt“, erklärt Andrea Rossi, der zusammen mit Zacharias Kontarakis die Studie federführend durchgeführt hat. Schaltet man hingegen durch einen genetischen Eingriff das Gen selbst aus, beeinträchtigt dies das Wachstum der Blutgefäße nicht.
Zunächst schlossen die Forscher in einem Experiment aus, dass etwaige Nebenwirkungen des Knock-down-Wirkstoffes für die Störung der Gefäßentwicklung verantwortlich sind. Dazu verabreichten sie die Substanz Fischlarven, bei denen das egfl7-Gen bereits ausgeschaltet war. Trotz der Behandlung entwickelten sich die Larven weitestgehend normal.
„Da der Wirkstoff keine Störung im Gefäßwachstum verursacht, haben wir einen anderen Mechanismus vermutet: Der Verlust eines Gens könnte kompensiert werden, indem ein anderes Gen die Aufgabe übernimmt“, sagte Kontarakis. „Wir haben deshalb nach Rettungsgenen gesucht, die in Tieren ohne funktionierendes egfl7-Gen verstärkt gebildet werden.“
Die Forscher verglichen daraufhin die gebildeten Boten-RNA-Moleküle und Proteine von Fischen ohne funktionstüchtiges egfl7-Gen mit denen normaler Tieren. Dabei stießen sie auf verschiedene mRNAs und deren Proteine, die in Fischen ohne egfl7 in größeren Mengen vorliegen. Ein Beispiel ist das Protein Emilin 3B. Werden Knock-down-Fischlarven mit Emilin 3B behandelt, in denen egfl7 zuvor blockiert worden war, entwickeln sich ihre Blutgefäße weitgehend normal. „Emilin 3B kann also den Ausfall von egfl7 kompensieren. In den egfl7-Knock-out-Fischen wird das Emilin verstärkt gebildet und kann so das Fehlen von egfl7 ausgleichen. In den Knock-down-Fischen geschieht das nicht“, erklärt Stainier.
Als nächstes will die Arbeitsgruppe analysieren, woher Gene „wissen“, dass ein anderes Gen ausgeschaltet wurde und wie sie dann den Verlust kompensieren. „Weltweit arbeiten viele Wissenschaftler daran, krank machende Gene auszuschalten und so Krankheiten zu behandeln. Für solche Therapien müssen wir unbedingt wissen, welche Auswirkungen der Verlust oder die Blockade eines Gens haben können. Zudem zeigt unsere Studie die Stärke eines Vergleichs zwischen Knock-down- und Knock-out-Daten, um modifizierende Gene zu identifizieren“, sagt Stainier. Dabei handelt es sich nach seiner Meinung um eine der großen Herausforderungen im Bereich der Humangenetik.
Max-Planck-Gesellschaft, 16. Juli 2015
Originalpublikation:
Andrea Rossi, Zacharias Kontarakis, Claudia Gerri, Hendrik Nolte, Soraya Hölper, Marcus Krüger & Didier Y. R. Stainier. Genetic compensation induced by deleterious mutations but not gene knockdowns. Nature; 13 July, 2015 doi:10.1038/nature14580

